佛山大學醫(yī)學部唐冬生團隊發(fā)布CRISPR/Cas9脫靶預測模型CCLMoff
發(fā)布時間:2025-06-13
CRISPR/Cas9系統(tǒng)為遺傳病基因治療提供了革命性工具,但同時會引起潛在的基因脫靶效應,已成為制約臨床轉化的核心障礙。現有脫靶預測工具受限于訓練數據規(guī)模和特定檢測平臺依賴,對未知序列的泛化能力不足。為解決這一領域痛點,近日佛山大學唐冬生教授團隊與深圳市衛(wèi)生健康發(fā)展研究和數據管理中心趙靚團隊聯合在《Communications Biology》(IF:5.2;中科院生物學一區(qū)Top期刊)發(fā)表創(chuàng)新成果,開發(fā)出首個基于RNA語言模型的CRISPR/Cas9通用脫靶預測模型:CCLMoff。
CCLMoff的核心創(chuàng)新在于其生物信息驅動的語言模型架構。研究團隊創(chuàng)造性地采用“問答框架“:將sgRNA序列視為“問題“,目標DNA序列經偽RNA化處理(胸腺嘧啶T→尿嘧啶U轉換)后作為“答案“。利用預訓練模型RNA-FM(基于RNAcentral數據庫中2300萬條RNA序列訓練)初始化12層Transformer編碼器,通過[SEP]標記分隔雙序列輸入,最終提取[CLS]標記的隱藏狀態(tài)經多層感知器來預測脫靶概率(圖1)。
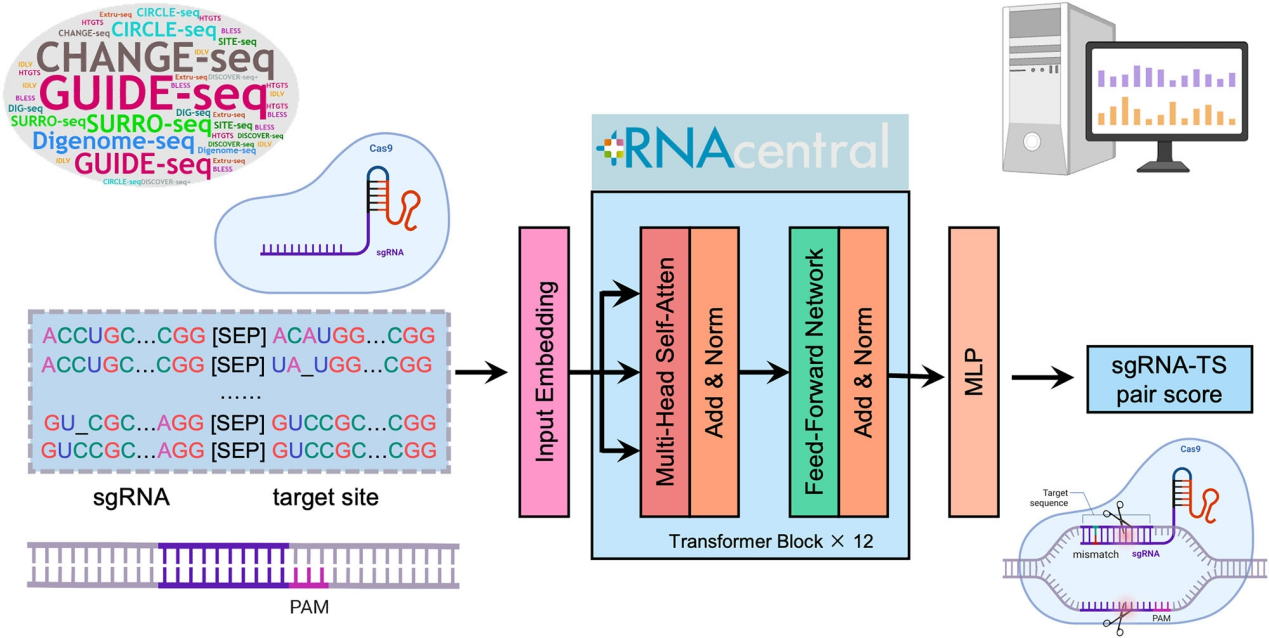
圖1 . CCLMoff模型架構
CCLMoff模型基于直接學習sgRNA與DNA相互作用模式進行思考,在平衡準確率、AUROC和AUPRC等多項評價指標上,都顯著優(yōu)于CRISPR-Net、Cas-OFFinder等現有模型。該模型還能突破傳統(tǒng)sgRNA 20nt的長度限制,對19/21nt非常規(guī)sgRNA的脫靶預測也同樣高效,泛化能力顯著增強。CCLMoff為克服脫靶效應這一CRISPR/Cas9應用核心難題提供了精準、泛化的AI解決方案,有望推動該領域研究進程。
本研究由國家重點研發(fā)計劃((2021YFA0805901)、國家自然科學基金(82070199)、廣東省基礎與應用基礎研究基金(2021A1515220078)和廣東省重點領域研發(fā)計劃(2022B0202110002)等項目資助完成。佛山大學為第一署名單位,佛山大學唐冬生教授、朱向星副教授和深圳市衛(wèi)生健康發(fā)展研究和數據管理中心趙靚博士為共同通訊作者。
撰稿人:朱向星
初審人:郭嘉亮
終審人:劉小輝







